最新发布第2页
Nature Methods|可解释机器学习在计算生物学中的应用与陷阱
在计算生物学和生物信息学领域,机器学习技术正迅速改变着我们对生物系统的研究方式。然而,随着模型复杂度的增加,如何解释这些模型的行为并从中提取生物学意义,成为了一个亟待解决的问题。 ...
ProtChat:融合大语言模型与蛋白质语言模型的自动化蛋白质分析工具
近年来,大语言模型(LLMs)在自然语言处理领域取得了巨大进展,极大地提升了人机交互的效率和精准度。而在计算生物学中,蛋白质序列被类比为自然语言,基于此的蛋白质大语言模型(PLLMs)也应...
深度学习与大语言模型开源书单
下面是一些开源的深度学习与大语言模型教程书籍,可以从 GitHub 免费下载。 神经网络与深度学习 链接: **https://nndl.github.io/ 介绍: 系统地整理了神经网络和深度学习的知识体系。鉴于深度...
第三方Docker安装与DockerHub镜像
如果因为网络问题无法下载安装 Docker,无法使用 DockerHub 镜像,可以使用下面的方法解决。 第三方 Docker 安装镜像 使用脚本镜像一键安装 Docker: sudo curl -fsSL https://gitee.com/tech-s...
生物信息学家、计算机科学家和遗传学家,谁主导开发的生物信息学工具更好?
新西兰奥塔哥大学高级讲师(Senior Lecturer ) Paul P. Gardner 在 bioRxiv 上发表了一篇名为《A Bioinformatician, Computer Scientist, and Geneticist lead bioinformatic tool development...
Ubuntu怎样查询和挂载未分区的硬盘
在Ubuntu上挂载一个未分区的硬盘需要几个步骤,包括分区、格式化和挂载。以下是详细的操作步骤: 1. 确认硬盘 首先,需要确认未分区的硬盘设备名。可以使用以下命令列出所有硬盘和分区: sudo f...
Ubuntu怎么查询和持久化挂载硬盘
在 Ubuntu 中,可以使用以下步骤来查询和持久化挂载硬盘: 查询硬盘信息 使用 lsblk 命令: lsblk 命令会列出所有的块设备(包括硬盘、分区等): lsblk 使用 fdisk 命令: fdisk 命令可以显示...
一文搞懂易混淆亲缘关系统计量(血缘系数、亲缘系数、近交系数)
背景 血缘系数、亲缘系数、近交系数等表征亲缘关系的统计量,在遗传学中经常使用。然而,这些概念很容易混淆。下面是几个概念的区别。注意的是,这些名词大多由英文翻译而来,而不同地方的中文...
Docker一键式脚本安装
通过脚本从 stable channel 自动安装稳定版: curl -fsSL https://get.docker.com -o get-docker.sh sh get-docker.sh 也可以选择 test channel 安装预发布版本(alpha, beta, rc)等: curl -f...
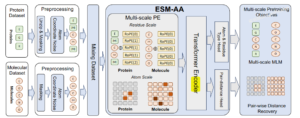
ICML 2024|用于统一分子建模的多尺度蛋白质语言模型ESM-AA
多模态蛋白质语言模型是一个新兴的方向。 《Multi-scale Protein Language Model for Unified Molecular Modeling》是一篇发表在ICML 2024的文献,作者是 Kangjie Zheng、Siyu Long、Tianyu Lu...
CVPR 2014| BioCLIP:生命之树的视觉基础模型
BioCLIP 是一个利用图像和结构化生物知识回答生物学问题的多模态大模型。通过在生命之树(生物学分类)上训练模型,增强了对自然界层次结构的理解,具有强大的可泛化性。文章还创建了 TreeOfLif...
Genome Biology|常用计算工具会产生相互矛盾和过于乐观的AUPRC值
在生物医学和生物信息学领域,PRC 和 AUPRC 有着非常广泛的应用。然而,常用的计算工具本身存在的问题可能会被研究人员忽略,从而可能导致对结果解读的偏差。2024 年 5 月,香港中文大学曹沁研...